

ChIP-seq 与 ChIP 实验的区别,一篇文章讲清楚
 2025-07-08 16:52:10
2025-07-08 16:52:10
来源/作者:普拉特泽-生物医学整体课题外包平台
普拉特泽生物为广大生命科学研究人员提供ChIP-seq 与 ChIP 实验,可根据实验方案的要求选择不同的检测方法,本文就跟大家一起继续探究ChIP-seq 与 ChIP 实验的区别,更加清楚明了两者的差异。
染色质免疫沉淀(ChIP)技术是研究蛋白质-DNA相互作用的核心方法,而ChIP-seq(ChIP结合高通量测序)是其高通量升级版本。两者在实验设计、数据产出和应用范围上存在显著差异。本文将系统比较ChIP与ChIP-seq的技术原理、实验流程、数据解析及适用场景~帮助大家更加了解清楚~
一、核心概念与原理对比
1. ChIP(染色质免疫沉淀)
原理:通过抗体富集特定蛋白结合的DNA片段,采用qPCR或微阵列(ChIP-chip)检测目标序列。
检测范围:
靶向分析(已知基因组位点,如启动子、增强子)
低通量(每次实验检测≤10个位点)
2. ChIP-seq(染色质免疫沉淀测序)
原理:将ChIP富集的DNA进行高通量测序,全基因组范围内定位蛋白结合位点。
检测范围:
全基因组覆盖(可发现未知结合位点)
单碱基分辨率(精确到1bp)
技术对比表
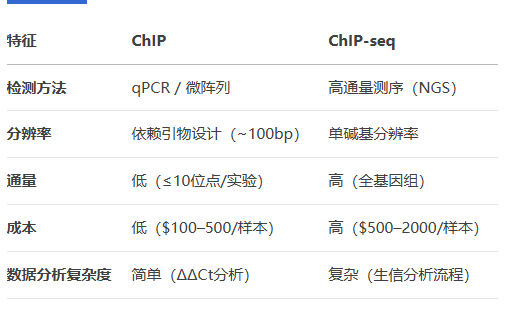
二、实验流程关键差异
1. 样本制备阶段
• 共同步骤:
◦ 甲醛交联(X-ChIP)或天然处理(N-ChIP)
◦ 染色质片段化(超声/MNase)
◦ 抗体免疫沉淀(IP)
• ChIP-seq特有优化:
◦ 片段大小选择:需严格控制在200–300bp(Illumina测序兼容)
◦ DNA末端修复:补平DNA末端并加A尾(适配体连接准备)
◦ 文库构建:需PCR扩增并纯化(推荐使用NEBNext® Ultra™ II试剂盒)
2. 检测与分析阶段
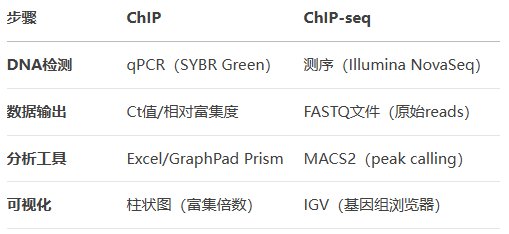
三、应用场景对比
1. ChIP的典型应用
• 验证性实验:
◦ 已知转录因子(如p53)在特定启动子的结合
◦ 组蛋白修饰(H3K27me3)在基因启动子的富集
• 低预算项目:
◦ 实验室初步筛选候选位点
2. ChIP-seq的核心优势
• 发现性研究:
◦ 全基因组转录因子结合位点鉴定(如CTCF motif分析)
◦ 新型增强子/沉默子识别
• 高分辨率分析:
◦ 单碱基突变对蛋白结合的影响(如癌症突变体研究)
◦ 差异结合分析(如治疗前后组蛋白修饰变化)
技术选择决策树
是否需要全基因组数据?
├── 是 → 选择ChIP-seq
└── 否 → 是否仅验证已知位点?
├── 是 → 选择ChIP-qPCR
└── 否 → 考虑ChIP-chip(微阵列)
四、数据深度与成本权衡
1. 数据产出对比

五、前沿进展与替代技术
1. ChIP-seq的技术升级
CUT&Tag:
无需交联,灵敏度提高10倍(适用于单细胞)
替代传统ChIP-seq的低样本量场景
Multi-omics整合:
ChIP-seq + ATAC-seq(开放染色质共分析)
2. ChIP的替代方案
▲DNA亲和纯化(DAP-seq):
适用于体外研究DNA结合蛋白
▲CUT&RUN:
在完整细胞中定位蛋白-DNA相互作用
六、结论
ChIP vs ChIP-seq 选择指南
选择ChIP-qPCR如果:
仅需验证少量已知位点
预算有限或无需全基因组数据
选择ChIP-seq如果:
需发现新结合位点或全基因组图谱
要求单碱基分辨率(如SNP影响分析)
普拉特泽,坚持提供真实、完整、唯一原始数据、原始图片。
普拉特泽生物,自有专业动物房、造模实验室,配套设备齐全,技术团队成熟,熟练掌握各类动物模型构建,更有细胞、分子、病理平台,一站式解决后续相关实验检测。请点击分子实验平台了解详情。

