

Taqman探针法检测SNP分型详解
 2022-08-04 15:12:43
2022-08-04 15:12:43
来源/作者:普拉特泽-生物医学整体课题外包平台
大家好,今天普拉特泽生物跟大家一起来学习探针法检测SNP的详细操作步骤,SNP全称单核苷酸多态性,是指在基因组上单个核苷酸的变异,变异类型包括转换、颠换、缺失和插入。普拉特泽——表达检测平台专业承接SNP检测代做外包服务与生物医学细胞实验技术培训,铸就你基础医学科研的金身。那现在我们来一点一点看Taqman探针法检测SNP的详细内容!
Taqman探针法将样品与反应组分在封闭的体系中进行简单混合,通过检测PCR反应过程中的荧光变化,可以实时检测SNP位点的基因分型。由于无需任何PCR之后的操作,这样就避免了PCR之后的人力成本与PCR后处理过程中的交叉污染。由于PCR反应的灵敏性,这种方法需要的样本含量极少。同时,随着日益成熟的荧光定量PCR仪,可以在一个反应板中进行大批量样本的同时检测。
Taqman探针法检测SNP的原理是:利用Taq DNA聚合酶的5-3核酸外切酶活力对探针本身进行酶切。Taqman探针的两端分别标记有荧光基团和对应的淬灭基团,当探针完整时,oligo相当于一条锁链将荧光基团和淬灭基团紧紧拴在一起,导致荧光基团发出的光被淬灭基团吸收。当探针被Taq酶切断之后,荧光基团得以释放从而导致荧光值上升,荧光值的变化会被荧光定量PCR仪或者其他检测仪器记录下来。
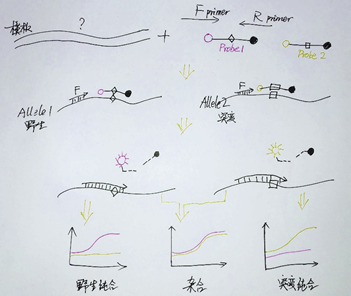
对于SNP分型检测,体系中会含有针对不同基因型的探针(如下图Probe1和Probe2),两种探针5端通过标记不同的荧光基团进行区分(通常使用的是FAM和VIC)。他们的序列中包含匹配不同基因型的碱基,碱基错配将导致探针与模板的结合能力以及被切割的概率大大降低。因此,当基因型为纯合子时,只会检测到单独一种荧光信号,而对于杂合子,两种荧光信号都将被检测到。
那有哪些因素会影响到探针对SNP分型效果呢?
首先,探针中错配的碱基需要对整条探针与模板的结合能力有比较大的影响,即含有错配的探针Tm值需比正常探针低得多。这对探针的长度及SNP位点在探针中的位置有较高的要求。
第二,由于分型探针是在同一个反应体系中,因此不同的探针与模板的结合是一种竞争状态,这就要求不同的分型探针与模板结合的位置是需要重叠的,这样才能保证一个模板上只结合一种探针。
第三,Taqman探针的水解是先解离一部分,然后再水解。Taq酶的5’核酸外切酶活性实际上识别的是一个Y型结构,其游离的单链至少为1~3个核苷酸,此时要求错配的探针应当更倾向于从模板上解离下来,而不是继续留在模板上被Taq酶水解。
为了能得到最合适的实验结果,我们列出了探针设计的各种原则:
1. 避免5’端第一个核苷酸为G,鸟嘌呤核苷酸对临近的荧光基团有明显的淬灭作用,导致结果信号值低。
2. 探针的Tm值需要比引物的Tm值高5~8°C,通常情况下为65~67°C
3. 为了增加探针的单碱基区分度,探针长度不宜过长,但同时MGB探针也不宜短于13个核苷酸。
4. 避免连续相同碱基的出现,尤其是连续的G,多余4个连续的G存在时,探针容易形成G四联体或其他高级结构,不利于其与模板的结合。
5. 区分SNP的碱基应当位于探针中间1/3的位置去(将探针平分三段,SNP位于中间那段)。如果这段位置不合适,设计不出优秀的探针,可以将SNP位置向3’端挪动,以更加靠近MGB基团。不要将SNP分型位点安排到最后的三个核苷酸中。
6. 由于G核苷酸形成高级结构及对荧光团的淬灭特性,探针的G残基数量不宜超过C的数量,如果有此情况,建议将探针设计在互补链上。
好啦,那关于Taqman探针法检测SNP分型咱们就讲解到这里啦,如果您还有其他的问题或者有SNP检测代做的需求欢迎随时官网留言,我们下期见~

